Tabella dei Contenuti
In un mondo sempre più guidato dai dati, l’apprendimento automatico si rivela uno strumento essenziale per estrarre valore dall’enorme quantità di informazioni disponibili. Tuttavia, affrontare problemi complessi richiede spesso approcci innovativi, in grado di andare oltre le divisioni tradizionali tra apprendimento supervisionato e non supervisionato.
Tradizionalmente, l’apprendimento supervisionato si concentra sulla costruzione di modelli che prevedono un’etichetta target utilizzando dati etichettati, mentre l’apprendimento non supervisionato è orientato all’identificazione di pattern nascosti nei dati privi di etichette.
Tuttavia, in scenari complessi e ricchi di variabilità la distinzione rigida tra supervisionato e non supervisionato può limitare le potenzialità predittive e interpretative. Combinare questi approcci rappresenta un’opportunità per migliorare l’accuratezza dei modelli e per estrarre informazioni più approfondite, specie in dataset scarsamente strutturati o con pattern difficili da rilevare.
- un approccio pratico e replicabile per integrare tecniche supervisionate e non
- utilizzare algoritmi supervisionati insieme a quelli di clustering
- analizzare e riportare risultati dei modelli
Perché combinare apprendimento supervisionato e non supervisionato?
In contesti pratici, i dati raramente si presentano in una forma completamente strutturata o priva di rumore. L’apprendimento supervisionato, pur potente, può essere limitato dalla qualità e dalla quantità di dati disponibili, soprattutto quando i pattern sottostanti non sono immediatamente evidenti. Dall’altro lato, l’apprendimento non supervisionato eccelle nell’identificare relazioni nascoste tra le variabili, ma non fornisce un output diretto per problemi di classificazione o regressione.
La sinergia tra i due approcci offre vantaggi significativi. I metodi non supervisionati possono, ad esempio, raggruppare dati apparentemente eterogenei in cluster interpretabili, che a loro volta possono essere utilizzati come feature aggiuntive per un modello supervisionato. Questo arricchimento del dataset migliora non solo la performance del modello, ma anche la sua robustezza, rendendolo più resiliente a variazioni nei dati.
Definizione di apprendimento supervisionato e non supervisionato
L’apprendimento automatico è uno strumento fondamentale della data science moderna, utilizzato per estrarre valore dai dati e costruire modelli predittivi. All’interno di questo vasto campo, le tecniche di apprendimento supervisionato e non supervisionato rappresentano i pilastri fondamentali. Nonostante siano spesso utilizzate separatamente, integrarli può amplificare le capacità analitiche e migliorare i risultati.
Cos’è l’apprendimento supervisionato?
L'apprendimento supervisionato è una branca dell'intelligenza artificiale e del machine learning (ML) che si occupa di insegnare ai modelli a fare previsioni o classificazioni basate su un dataset etichettato. Ogni esempio nel dataset contiene una serie di caratteristiche di input (variabili indipendenti) e un'etichetta associata (variabile target) che rappresenta l'output desiderato.
Cos'è un'etichetta?
Un'etichetta è il valore associato a ciascuna osservazione in un dataset. Rappresenta l'output corretto o il risultato atteso che il modello dovrebbe imparare a prevedere. Le etichette sono fondamentali nei problemi di apprendimento supervisionato, in cui i dati di input sono accompagnati da una risposta conosciuta per guidare l'addestramento del modello.
Esempio:
- In un problema di classificazione delle immagini, l'etichetta potrebbe indicare la categoria dell'oggetto nell'immagine (ad esempio, "gatto" o "cane").
- In un problema di regressione, l'etichetta potrebbe essere un valore numerico, come il prezzo di una casa basato su caratteristiche come dimensioni e posizione.
Caratteristiche delle etichette:
- Tipologia:
- Categoriali per problemi di classificazione (es. "poco", "medio", "tanto").
- Numeriche per problemi di regressione (es. età, peso o altezza).
L’etichetta è cruciale perché guida l'apprendimento del modello. Errori o inaffidabilità nelle etichette possono compromettere le performance del sistema.
Caratteristiche principali dell’apprendimento supervisionato
- Richiede dati con etichette: Ogni esempio deve includere sia gli input che le etichette. La qualità delle etichette è determinante per l’efficacia del modello.
- Obiettivo: Minimizzare la differenza (errore) tra le previsioni del modello e le etichette reali. Per fare ciò, si ottimizza una funzione di perdita come:
- Cross-Entropy Loss per la classificazione.
- Mean Squared Error per la regressione.
- Applicazioni:
- Classificazione: Distinzione tra categorie (es. adulti con reddito ≤50K o >50K);
- Regressione: Predizione di ì un valore continuo (es. stima dell'esatta temperatura di un paziente).
Algoritmi comuni:
- XGBoost e Random Forest: Utili per problemi strutturati con variabili numeriche o categoriali.
- Reti Neurali: Ideali per dati complessi come immagini o testo.
Cos’è l’apprendimento non supervisionato?
L’apprendimento non supervisionato è una branca del machine learning che opera su dataset privi di etichette, ossia senza informazioni esplicite sulle categorie o valori target. L’obiettivo principale è scoprire pattern nascosti, strutture latenti, o relazioni sottostanti tra i dati, in modo da estrarre valore informativo anche in assenza di supervisione esplicita.
Caratteristiche principali:
- Assenza di etichette: Non richiede una variabile target per guidare l’apprendimento.
- Induttività: Gli algoritmi cercano pattern nei dati basandosi su regole e ipotesi predefinite (ad esempio similarità o densità).
- Obiettivi principali:
- Clustering: Suddivisione delle osservazioni in gruppi coerenti.
- Riduzione della dimensionalità: Semplificazione dei dati mantenendo il massimo contenuto informativo.
- Individuazione di anomalie: Rilevazione di punti che si discostano dalla struttura attesa.
- Applicazioni: Segmentazione del mercato, compressione dei dati, analisi dei comportamenti, esplorazione iniziale dei dataset.
Algoritmi comuni:
- K-Means: Suddivide i dati in gruppi con caratteristiche simili, minimizzando la distanza tra i punti e i centroidi.
- DBSCAN: Identifica cluster di forma arbitraria, considerando densità e vicinanza.
- Clustering Gerarchico: Crea una struttura ad albero che rappresenta relazioni gerarchiche tra osservazioni.
Se vuoi leggere un articolo introduttivo al machine learning, puoi cliccare sul link qui sotto 👇

Vantaggi dell’integrazione
L’integrazione tra apprendimento supervisionato e non supervisionato consente di combinare la potenza predittiva dei modelli supervisionati con la capacità dei metodi non supervisionati di identificare pattern nascosti nei dati. Questo approccio può arricchire il dataset con nuove feature e migliorare significativamente le performance dei modelli.
Benefici principali:
- Identificazione di pattern latenti: I cluster derivati da tecniche non supervisionate possono rappresentare gruppi con caratteristiche comuni, spesso invisibili a un’analisi diretta.
- Aggiunta di nuove feature: Etichette di cluster, distanze dai centroidi e indicatori di densità possono diventare preziose variabili predittive.
- Maggiore robustezza: Modelli ibridi tendono ad essere più resistenti ai problemi di overfitting o a variazioni nei dati.
Il dataset
Il dataset Wine Quality - Red è un insieme di dati ampiamente utilizzato in ambito di machine learning, in particolare per problemi di regressione e classificazione multiclasse.
Questo dataset viene spesso impiegato per prevedere la qualità del vino rosso, valutata su una scala da 0 a 10, sulla base delle sue caratteristiche fisico-chimiche. È una risorsa ideale per introdurre tecniche di data preprocessing, esplorazione dei dati e modellazione predittiva, e per testare algoritmi come regressione lineare, alberi decisionali, support vector machines e reti neurali.
Variabili del dataset
Il dataset contiene 11 variabili indipendenti e una variabile target. Di seguito, un elenco dettagliato delle variabili presenti:
- fixed acidity: Acido tartarico nel vino, misurato in g/dm³ (variabile continua).
- volatile acidity: Acido acetico, responsabile dell’acidità volatile, misurato in g/dm³ (variabile continua).
- citric acid: Acido citrico, associato alla freschezza del vino, misurato in g/dm³ (variabile continua).
- residual sugar: Zuccheri residui non fermentati, misurati in g/dm³ (variabile continua).
- chlorides: Contenuto di cloruro di sodio, indicativo della salinità, misurato in g/dm³ (variabile continua).
- free sulfur dioxide: Diossido di zolfo libero, utile per prevenire l’ossidazione, misurato in mg/L (variabile continua).
- total sulfur dioxide: Totale diossido di zolfo, una combinazione di libero e legato, misurato in mg/L (variabile continua).
- density: Densità del vino, misurata in g/cm³ (variabile continua).
- pH: Livello di acidità del vino, scala logaritmica (variabile continua).
- sulphates: Solfati, associati alla conservazione del vino, misurati in g/dm³ (variabile continua).
- alcohol: Percentuale di alcol presente nel vino (variabile continua).
- quality: Variabile target, rappresenta la qualità del vino, valutata da esperti su una scala da 0 a 10 (variabile categorica).
Questo dataset rappresenta un esempio pratico di come le proprietà chimiche di un prodotto possano essere correlate alle sue caratteristiche sensoriali, offrendo un caso d'uso interessante sia per la ricerca accademica che per applicazioni industriali.
Libreria Python per ottenere il dataset
In Python, il dataset Wine Quality Red del repository UCI è facilmente accessibile tramite librerie come pandas o pacchetti esterni come OpenML, che forniscono dataset pronti per l'uso.
Sebbene scikit-learn offra strumenti integrati per caricare dataset di esempio come load_iris, il dataset Wine Quality Red è generalmente disponibile in formato CSV. Tuttavia, OpenML consente di caricare il dataset direttamente nel codice senza doverlo scaricare manualmente.
Ecco un esempio per ottenere il dataset utilizzando OpenML:
import pandas as pd
# URL of the dataset from the UCI repository
url = "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-red.csv"
# Loading the dataset with ';' as the delimiter
df = pd.read_csv(url, sep=";")
# Initial exploration
print(df.head())
>>>
fixed acidity volatile acidity citric acid residual sugar chlorides \
0 7.4 0.70 0.00 1.9 0.076
1 7.8 0.88 0.00 2.6 0.098
2 7.8 0.76 0.04 2.3 0.092
3 11.2 0.28 0.56 1.9 0.075
4 7.4 0.70 0.00 1.9 0.076
free sulfur dioxide total sulfur dioxide density pH sulphates \
0 11.0 34.0 0.9978 3.51 0.56
1 25.0 67.0 0.9968 3.20 0.68
2 15.0 54.0 0.9970 3.26 0.65
3 17.0 60.0 0.9980 3.16 0.58
4 11.0 34.0 0.9978 3.51 0.56
alcohol quality
0 9.4 5
1 9.8 5
2 9.8 5
3 9.8 6
4 9.4 5
Con questa soluzione, si ottiene il dataset già formattato come DataFrame di Pandas, rendendo semplice la manipolazione e l'analisi.
Pre-elaborazione dei dati
La pre-elaborazione dei dati rappresenta un passo cruciale per il successo di qualsiasi modello di apprendimento automatico, supervisionato o non supervisionato. Utilizzando il Wine quality Red Dataset come esempio, esploreremo i passaggi fondamentali per pulire e trasformare i dati in modo che siano pronti per l’analisi.
1. Esplorazione Iniziale del Dataset
Prima di iniziare con la pre-elaborazione, è necessario comprendere la struttura del dataset:
- Identificare il numero di osservazioni e feature.
- Controllare la presenza di valori mancanti o inconsistenti.
- Esaminare la distribuzione delle variabili per evidenziare squilibri o outlier.
# Print some essential information
print(f"Dimensioni del dataset: {df.shape}")
print("\nTipi di dati:\n", df.dtypes)
print("\nValori mancanti:\n", df.isnull().sum())
>>>
Dimensioni del dataset: (1599, 12)
Tipi di dati:
fixed acidity float64
volatile acidity float64
citric acid float64
residual sugar float64
chlorides float64
free sulfur dioxide float64
total sulfur dioxide float64
density float64
pH float64
sulphates float64
alcohol float64
quality int64
dtype: object
Valori mancanti:
fixed acidity 0
volatile acidity 0
citric acid 0
residual sugar 0
chlorides 0
free sulfur dioxide 0
total sulfur dioxide 0
density 0
pH 0
sulphates 0
alcohol 0
quality 0
dtype: int64
2. Normalizzare le variabili numeriche
La normalizzazione è un processo fondamentale in cui i valori delle variabili numeriche vengono trasformati in modo da rientrare in una scala comune, solitamente mantenendo l'intervallo tra 0 e 1 o con media 0 e deviazione standard 1. Questo passaggio è cruciale per garantire che le diverse variabili siano confrontabili, indipendentemente dalle loro unità di misura o dai range di valori.
La normalizzazione è essenziale per molti algoritmi di machine learning, come il clustering, la regressione logistica e le reti neurali, poiché questi metodi sono sensibili alle differenze di scala tra le variabili.
from sklearn.preprocessing import MinMaxScaler
# Exclude 'quality' from normalization
columns_to_normalize = [col for col in df.columns if col != 'quality']
scaler = MinMaxScaler()
df[columns_to_normalize] = scaler.fit_transform(df[columns_to_normalize])
print("\nDataset dopo normalizzazione (escludendo 'quality'):\n", df.head())
>>>
Dataset dopo normalizzazione (escludendo 'quality'):
fixed acidity volatile acidity citric acid residual sugar chlorides \
0 0.247788 0.397260 0.00 0.068493 0.106845
1 0.283186 0.520548 0.00 0.116438 0.143573
2 0.283186 0.438356 0.04 0.095890 0.133556
3 0.584071 0.109589 0.56 0.068493 0.105175
4 0.247788 0.397260 0.00 0.068493 0.106845
free sulfur dioxide total sulfur dioxide density pH sulphates \
0 0.140845 0.098940 0.567548 0.606299 0.137725
1 0.338028 0.215548 0.494126 0.362205 0.209581
2 0.197183 0.169611 0.508811 0.409449 0.191617
3 0.225352 0.190813 0.582232 0.330709 0.149701
4 0.140845 0.098940 0.567548 0.606299 0.137725
alcohol quality
0 0.153846 5
1 0.215385 5
2 0.215385 5
3 0.215385 6
4 0.153846 5
5. Divisione in Set di Addestramento e Test
In un contesto di apprendimento non supervisionato, non è strettamente necessario dividere i dati in train e test set come si fa per i modelli supervisionati. Questo perché l'obiettivo dell'apprendimento non supervisionato non è predire un output specifico, ma piuttosto identificare pattern, strutture latenti o gruppi nei dati.
Tuttavia, ci sono alcune considerazioni pratiche che potrebbero richiedere una suddivisione dei dati anche in ambiti non supervisionati, in particolare quando l'apprendimento non supervisionato è integrato in un framework supervisionato.
Se il clustering viene utilizzato per creare feature latenti (es. etichette di cluster) da includere in un modello supervisionato, è necessario applicare il clustering solo sui dati di training. Questo per evitare un problema noto come data leakage, ovvero l'inclusione di informazioni dei dati di test durante la fase di training del modello supervisionato.
from sklearn.model_selection import train_test_split
# Separation of features and the target variable
X = df.drop('quality', axis=1)
y = df['quality']
# Splitting the data
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
print(f"\nDimensioni del set di addestramento: {X_train.shape}")
print(f"Dimensioni del set di test: {X_test.shape}")
>>>
Dimensioni del set di addestramento: (1279, 12)
Dimensioni del set di test: (320, 12)
Il dataset è stato suddiviso in due parti: un set di addestramento (train set) e un set di test (test set) in proporzione 80/20:
- 80% dei dati per il set di addestramento (
X_train,y_train), usato per costruire e ottimizzare il modello. - 20% dei dati per il set di test (
X_test,y_test), usato per valutare le performance del modello su dati non visti.
La funzione train_test_split garantisce che la divisione avvenga in modo casuale, ma grazie al parametro random_state, la procedura è riproducibile.
Apprendimento non supervisionato: Identificazione di gruppi latenti
L'integrazione di tecniche non supervisionate nell'analisi dei dati permette di scoprire pattern latenti e gruppi omogenei all'interno di un dataset. Nel contesto dell'Adult Income Dataset, l'obiettivo principale è identificare gruppi di individui con caratteristiche simili, utilizzando algoritmi di clustering come K-Means. Successivamente, queste informazioni saranno utilizzate per arricchire i modelli supervisionati con nuove feature latenti.
Obiettivi del clustering nel framework
- Identificazione di gruppi latenti: Ad esempio, individuare gruppi basati su età, livello di istruzione e ore lavorate settimanalmente.
- Arricchimento del dataset: Creare feature latenti, come l'etichetta del cluster e la distanza dal centroide, per migliorare le predizioni supervisionate.
Clustering con K-Means
Implementiamo il clustering utilizzando K-Means, scegliendo un valore di k ottimale basato sull’elbow method.
Il K-Means è un algoritmo di clustering non supervisionato che suddivide i dati in k gruppi o cluster. Inizia posizionando casualmente k centri (centroidi). Ogni punto dati viene assegnato al cluster con il centro più vicino, calcolando la distanza euclidea. Successivamente, i centroidi vengono aggiornati come media dei punti assegnati a ciascun cluster. Questo processo si ripete iterativamente finché i centroidi non si stabilizzano o la variazione è minima. Il risultato è una suddivisione dei dati che massimizza la similarità interna al cluster e minimizza la similarità tra cluster diversi, rendendo K-Means ideale per identificare strutture nei dati.
L’elbow method è una tecnica per determinare il numero ottimale di cluster (k) nel K-Means. Si calcola la somma delle distanze quadrate all’interno di ciascun cluster (intra-cluster variance) per valori crescenti di k. Questa misura, detta somma dei quadrati delle distanze (SSE), diminuisce all’aumentare di k poiché i cluster diventano più piccoli. Tuttavia, oltre un certo punto, la riduzione diventa marginale, formando un "gomito" nel grafico SSE vs k. Questo punto identifica k ottimale, bilanciando precisione del clustering e semplicità, evitando di sovra-segmentare i dati.
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score, accuracy_score
from sklearn.ensemble import RandomForestClassifier
import numpy as np
import matplotlib.pyplot as plt
# Automatically select the best number of clusters using silhouette score
sil_scores = []
k_range = range(2, 11)
for k in k_range:
kmeans = KMeans(n_clusters=k, random_state=42)
labels = kmeans.fit_predict(X_train)
sil_score = silhouette_score(X_train, labels)
sil_scores.append(sil_score)
# Find the best k based on silhouette score
best_k = k_range[np.argmax(sil_scores)]
print(f"Best number of clusters (k): {best_k}")
# Fit KMeans with the best number of clusters
kmeans = KMeans(n_clusters=best_k, random_state=42)
cluster_labels_train = kmeans.fit_predict(X_train)
cluster_labels_test = kmeans.predict(X_test)
# Compute inertia for elbow method
inertia = []
k_values = range(2, 11)
for k in k_values:
kmeans = KMeans(n_clusters=k, random_state=42)
kmeans.fit(X_train)
inertia.append(kmeans.inertia_)
# Optimal k for visualization
optimal_k = best_k
# Print the best number of clusters and cluster centroids
print(f"Optimal number of clusters: {best_k}")
print(f"Cluster centroids:\n{kmeans.cluster_centers_}")
# Elbow Method plot
plt.figure(figsize=(8, 5))
plt.plot(k_values, inertia, marker='o', label="Inertia")
plt.axvline(x=optimal_k, color='r', linestyle='--', label=f"Optimal k={optimal_k}")
plt.xlabel('Number of clusters (k)')
plt.ylabel('Inertia')
plt.title('Elbow Method to Determine Optimal k')
plt.legend()
plt.show()
>>>
Best number of clusters (k): 2
Optimal number of clusters: 2
Cluster centroids:
[[0.63358444 0.20038492 0.53396694 0.11850447 0.12687813 0.15038994
0.10104255 0.68025873 0.3053296 0.2202702 0.25246874]
[0.26104418 0.36445783 0.08445783 0.09117291 0.12121944 0.13855422
0.09380277 0.498909 0.48746166 0.15645817 0.20570487]
[0.36738807 0.29797911 0.36574257 0.24281161 0.13975438 0.4385023
0.34482035 0.60731161 0.41654323 0.21752535 0.22894136]
[0.50221239 0.18510274 0.5261 0.16732877 0.12951586 0.0884507
0.05250883 0.54478708 0.37212598 0.24197605 0.48692308]
[0.10831858 0.30246575 0.0936 0.08486301 0.08230384 0.21239437
0.15038869 0.23262849 0.66834646 0.18107784 0.65758974]
[0.35667139 0.27348066 0.32281768 0.09679861 0.13178502 0.15290639
0.13702829 0.5464421 0.42271719 0.19227843 0.22269443]
[0.30590598 0.14711454 0.39744681 0.08836102 0.10033271 0.20542403
0.1064331 0.35608357 0.44351371 0.22699282 0.50103655]
[0.3308965 0.28886242 0.52391304 0.0723645 0.57973434 0.22167789
0.21155323 0.50983209 0.23895926 0.61155949 0.15518395]
[0.19919959 0.37317861 0.06980892 0.10084199 0.1100454 0.22266081
0.09034233 0.38541111 0.56331812 0.17227964 0.40053895]
[0.25953029 0.29140323 0.19961538 0.09619775 0.11477462 0.35148068
0.26736205 0.4756815 0.45956996 0.15384615 0.21300131]]
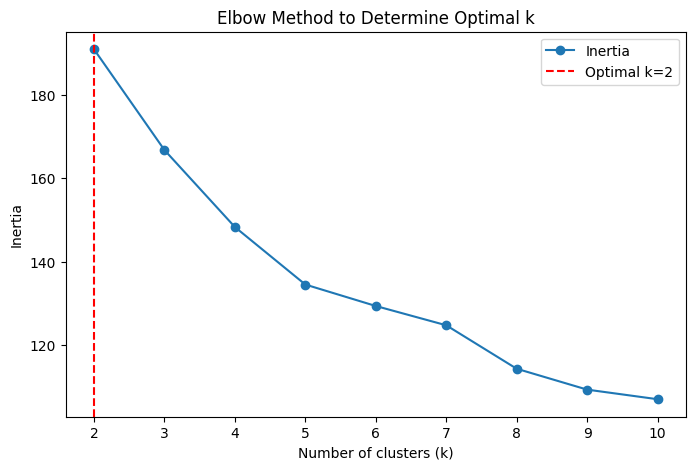
Per scegliere il valore massimo di k in un'analisi di clustering, è importante considerare alcune caratteristiche del dataset. Innanzitutto, la conoscenza del dominio gioca un ruolo fondamentale. Se si prevede che i dati siano suddivisi naturalmente in 3-5 gruppi, il range di k può essere ristretto a valori compresi tra 3 e 10.
Anche la dimensione del dataset influisce sulla scelta. Con dataset di piccole dimensioni, con meno di 100 osservazioni, è possibile testare un intervallo più ampio di valori di k, ad esempio da 1 a 10.
Esistono anche delle regole empiriche utili per determinare un limite superiore di k. Una di queste suggerisce che k non dovrebbe superare la metà del numero di osservazioni nel dataset (k ≤ n/2).
Per interpretare i risultati del K-Means con 2 cluster, possiamo analizzare i centroidi di ogni cluster rispetto alle variabili selezionate per il clustering. I centroidi rappresentano i "prototipi" dei gruppi identificati dall'algoritmo e forniscono una sintesi delle caratteristiche principali di ciascun cluster.
# Cluster Description
cluster_characteristics = X_train.groupby('Cluster').mean()
print("\nCaratteristiche medie per cluster:")
print(cluster_characteristics)
>>>
Caratteristiche medie per cluster:
fixed acidity volatile acidity citric acid residual sugar \
Cluster
0 0.451125 0.203460 0.464084 0.129712
1 0.245139 0.335163 0.139483 0.102059
chlorides free sulfur dioxide total sulfur dioxide density \
Cluster
0 0.143257 0.191686 0.132489 0.546822
1 0.116777 0.221901 0.151422 0.454317
pH sulphates alcohol
Cluster
0 0.379230 0.244869 0.346027
1 0.499317 0.164825 0.285804
Cluster 0: Vini equilibrati con acidità moderata e struttura robusta
- Fixed acidity: Valori medi superiori rispetto al Cluster 1, suggerendo una struttura più robusta e corposa.
- Volatile acidity: Inferiore rispetto al Cluster 1, indicando un'acidità volatile più bilanciata e un profilo aromatico più stabile.
- Citric acid: Valore medio più elevato, contribuendo a una sensazione di freschezza e complessità al palato.
- Residual sugar: Leggermente superiore rispetto al Cluster 1, indicando vini con un tocco di dolcezza residua.
- Chlorides: Valori medi più elevati, suggerendo un sapore leggermente salino o minerale.
- Free sulfur dioxide: Concentrazioni inferiori rispetto al Cluster 1, che possono contribuire a una percezione di maggiore naturalezza nel vino.
- Total sulfur dioxide: Inferiore rispetto al Cluster 1, suggerendo una minore aggiunta di conservanti.
- Density: Più alta rispetto al Cluster 1, coerente con una maggiore concentrazione di zuccheri e sostanze estrattive.
- pH: Valore medio inferiore, suggerendo un'acidità più accentuata e vivace.
- Sulphates: Valori più alti, contribuendo a una struttura solida e una maggiore persistenza aromatica.
- Alcohol: Gradazione alcolica media più alta, indicando vini più corposi e intensi.
Cluster 1: Vini leggeri e freschi con profilo aromatico semplice
- Fixed acidity: Valore medio inferiore, suggerendo vini con una struttura più delicata.
- Volatile acidity: Più elevata rispetto al Cluster 0, che può contribuire a note aromatiche più accentuate, ma anche a un'acidità percepita più pronunciata.
- Citric acid: Valore medio molto basso, riducendo la sensazione di freschezza.
- Residual sugar: Inferiore rispetto al Cluster 0, suggerendo vini più secchi.
- Chlorides: Valore medio più basso, contribuendo a un profilo più fresco e meno minerale.
- Free sulfur dioxide: Leggermente superiore rispetto al Cluster 0, migliorando la stabilità e la longevità del vino.
- Total sulfur dioxide: Più alto rispetto al Cluster 0, suggerendo una maggiore protezione contro ossidazione e contaminazioni.
- Density: Inferiore rispetto al Cluster 0, coerente con vini più leggeri e meno concentrati.
- pH: Valore medio più alto, suggerendo un’acidità più morbida.
- Sulphates: Inferiore rispetto al Cluster 0, riducendo la complessità strutturale del vino.
- Alcohol: Gradazione alcolica media inferiore, indicando vini più leggeri e meno corposi.
Apprendimento supervisionato: Random Forest
Implementiamo il clustering utilizzando il Random Forest, sfruttando l'affinità basata sulle distanze per costruire una matrice di similarità tra osservazioni.
Il Random Forest è un algoritmo di apprendimento automatico supervisionato utilizzato sia per la classificazione che per la regressione, ma può essere adattato al clustering non supervisionato. Funziona costruendo una moltitudine di alberi decisionali durante l'addestramento. Ogni albero è addestrato su un sottoinsieme casuale dei dati e delle variabili, aumentando la robustezza e riducendo il rischio di overfitting.
I parametri principali includono:
- Il numero di alberi nella foresta (n_estimators), che influisce sulla stabilità delle previsioni.
- La profondità massima degli alberi (max_depth), che regola la complessità del modello.
- La frazione di dati e variabili utilizzati per costruire ogni albero (max_features e max_samples).
Nel clustering, le distanze derivate dall'importanza delle variabili e dalla classificazione delle osservazioni vengono utilizzate per creare una matrice di similarità, permettendo di identificare strutture e gruppi nascosti nei dati. Questo approccio è particolarmente efficace con dati complessi e di alta dimensionalità.
# Baseline Random Forest classifier without clustering
rf = RandomForestClassifier(n_estimators=100, random_state=42)
rf.fit(X_train_full, y_train)
y_pred = rf.predict(X_test_full)
baseline_accuracy = accuracy_score(y_test, y_pred)
print(f"Baseline Accuracy: {baseline_accuracy:.4f}")
>>>
Baseline Accuracy: 0.6500
Per proseguire con l'analisi, integrando il framework proposto nello script, estraiamo dai risultati del K-Means informazioni utili per l'ingegneria delle feature e migliorare i modelli supervisionati, come suggerito nel documento. Le informazioni includeranno:
- Etichetta del cluster: da aggiungere al dataset come una nuova feature.
- Distanze dai centroidi: per fornire informazioni sulla prossimità di ciascun punto ai diversi gruppi.
# Train Random Forest classifier on the new dataset with cluster labels
rf_new = RandomForestClassifier(n_estimators=100, random_state=42)
rf_new.fit(X_train_new, y_train)
y_pred_new = rf_new.predict(X_test_new)
new_accuracy = accuracy_score(y_test, y_pred_new)
print(f"New Accuracy with Clustering: {new_accuracy:.4f}")
# Calculate the percentage improvement
improvement = (new_accuracy - baseline_accuracy) / baseline_accuracy * 100
print(f"Improvement Percentage: {improvement:.2f}%")
>>>
New Accuracy with Clustering: 0.6750
Improvement Percentage: 3.85%
L'integrazione dei cluster come etichette nel dataset ha portato a un'accuratezza del modello di classificazione Random Forest pari a 0.6750, evidenziando un miglioramento rispetto al modello di base del 3.85%. Questo risultato suggerisce che l'aggiunta di informazioni derivate dal clustering ha contribuito a migliorare la capacità del modello di distinguere le classi nei dati di test.
Tuttavia, il miglioramento percentuale, seppur positivo, è relativamente contenuto. Questo potrebbe indicare che i cluster forniscono informazioni aggiuntive utili, ma non significativamente determinanti per la classificazione.
È possibile che i cluster catturino solo una parte della struttura informativa latente nei dati, o che i dati di input presentino una correlazione relativamente debole con le etichette target originali.
Conclusioni
Nel panorama in continua evoluzione della data science e del machine learning, la capacità di innovare e integrare approcci diversi è essenziale per affrontare la complessità dei problemi reali. La combinazione di apprendimento supervisionato e non supervisionato rappresenta un passo fondamentale in questa direzione. Attraverso l'esplorazione delle loro sinergie, si aprono nuovi orizzonti per migliorare non solo l'accuratezza delle previsioni, ma anche la comprensione dei dati e delle relazioni nascoste al loro interno.
La fusione dei due paradigmi consente di superare i limiti intrinseci di ciascun approccio. L'apprendimento non supervisionato, grazie alla sua capacità di scoprire pattern nascosti, può generare nuove feature, raggruppare dati eterogenei e fornire insight interpretativi che arricchiscono i modelli supervisionati. Al contrario, l'apprendimento supervisionato sfrutta queste nuove informazioni per migliorare la precisione e la generalizzazione, dando luogo a modelli predittivi più robusti.
Per implementare questa combinazione, è necessario un approccio strutturato che parta dalla comprensione dei dati disponibili e delle loro limitazioni. Un framework tipico prevede:
- Analisi Esplorativa: Identificare pattern nascosti con algoritmi come K-Means, DBSCAN o clustering gerarchico.
- Integrazione dei Risultati: Utilizzare le etichette o altre informazioni derivate come feature per un modello supervisionato.
- Valutazione Iterativa: Misurare l'impatto delle nuove feature sulla precisione del modello, ottimizzando i parametri di entrambi gli approcci.
Questo processo richiede attenzione alla qualità dei dati e all'equilibrio tra complessità del modello e interpretabilità. Integrare apprendimento supervisionato e non supervisionato non è privo di ostacoli.
Tra le principali sfide:
- Scelta dei Parametri: Determinare il numero di cluster ottimale è cruciale. Metodi come l'Elbow Method o il Silhouette Score offrono indicazioni, ma richiedono spesso un'analisi manuale.
- Overfitting: L'aggiunta di troppe feature basate su clustering può portare a modelli eccessivamente complessi e poco generalizzabili.
- Interpretabilità: Mentre i metodi non supervisionati forniscono insight, integrarli in modelli supervisionati può ridurre la trasparenza complessiva.
Unire apprendimento supervisionato e non supervisionato rappresenta un'opportunità unica per massimizzare il valore dei dati. Questo approccio richiede competenze tecniche avanzate, ma offre risultati straordinari in termini di performance e insight. Adottare un framework ibrido è una strategia vincente per affrontare la crescente complessità dei problemi reali, aprendo nuove prospettive per la data science e il machine learning.
Seguire questo percorso non significa solo migliorare i modelli, ma anche contribuire a una comprensione più profonda del mondo basata sui dati, trasformando sfide in opportunità.







Commenti dalla community